TopHat/Cufflinks/CummeRbund使用介绍
TopHat、Cufflinks和cummeRbund,被称为处理RNA-seq数据的“燕尾服(tuxedo)”。TopHat负责RNA-seq的reads映射比对到基因组,并且自动识别mRNA“内含子-外显子”剪切;Cufflinks擅长组装转录组和寻找差异表达基因(或转录起始位点TSS等);cummeRbund主攻数据可视化。
1. Tophat
简介:TopHat是快速将RNA测序片段“对应(mapping)”到基因组上的工具,优势在于处理外显子间的剪切。内部首先使用bowtie或bowtie2把RNA测序片段“比对(alignment)”到基因组,之后再分析和鉴定剪切连接区域。
平台:Mac OS/Linux
快速运行:
# 双端测序
$ tophat2 -p 8 -o human_1 \
--mate-inner-dist 165 --mate-std-dev 45 --no-mixed \
hg19 human_1.fastq.gz human_2.fastq.gz
# 单端测序
$ tophat2 -p 8 -o human_1 hg19 human_1.fastq.gz
重要参数解释:
-p:设置线程数,用于多核计算。-o:执行输入文件夹。-r/--mate-inner-dist:一对reads的间隔长度的期望平均值,默认值为50bp,建议添加。具体解释,参考RNA-seq差异表达分析工作流程和Tophat中-r/–mate-inner-dist参数。--mate-std-dev:一对reads的间隔长度分布的标准差,默认值为20bp,建议添加。-r/--mate-inner-dist和--mate-std-dev的估计方法参考确定TopHat中mate-inner-dist参数。--library-type:测序仪器和方法,默认为标准Illumina平台的
fr-unstranded。其他平台设置,详见TopHat说明文档、How to tell which library type to use (fr-firststrand or fr-secondstrand)?和链特异性转录组原理。如果分不清楚
fr-firststrand和fr-secondstrand,推荐两种方法:第一种用两个参数试运行一个有1M reads的小样本,之后比较junction.bed大小;第二种在两个双端测序文件(fastq.gz)中抽取一些reads,之后Blat到USCS genomes上观察。
--no-discordant:只对于paired reads,只报告concordant mappings。加入这个参数,tophat2在最后一步失败。也可以不加入这个参数,通过sam/bam文件第二列过滤discordant reads,方法参考过滤TopHat分析双端测序的输出。--no-mixed:只对于paired reads,只报告paired reads都成功map。TopHat默认不加这个参数,即如果对于一个read,如果没有找到alignment的concordant或者discordant mate,那么这一对read将分别寻找和报道各自的alignment。这个参数与--no-discordant不同,因为加上--no-mixed也可能报道discordant pairs(例如一对reads都成功alignment,但是方向或者之间距离不对)。-g/--max-multihits:对于多个map的reads,设定报告数目,默认数值为20。需要注意,尽管这个参数可以设定为1,也不能用于设定唯一map的read。因为某个read有可能map到基因组多个位点,当设定为1时,只会返回得分最高的情况。
后续操作:
TopHat2运行后查看
align_summary.txt获得比对结果。TopHat2会输出
accepted_hits.bam(接受map的reads文件)和unmapped.bam(没有map上的reads文件)。对于后者,使用基因组浏览器,如IGV或者UCSC Genome Browser大致看下是有无map,之后可以直接丢弃。过滤双端测序的TopHat结果,参考过滤TopHat分析双端测序的输出。
补充:
TopHat2运行时,可以将基因组的fasta格式文件(比如
hg19.fa),一起放在index的文件夹中(如果没有,Bowtie2先生成一个),可以节省运行时间。具体生成方法,参考:二代测序中的短序列比对。
2. Cufflinks
简介:Cufflinks是TopHat的下游工具,用于分析差异表达基因、差异转录起始位点、新基因和选择性剪切。一般可以分为三步:1. cufflinks对每个bam文件生成转录组;2. cuffmerge结合真实转录组和bam生成转录组,构建一个整合转录组;3. cuffdiff比较不同生物学样本,寻找差异表达基因。
平台:Mac OS/Linux
2.1 cufflinks 快速运行
$ cufflinks -p 8 -g hg19_ensembl.gtf -b hg19.fa -M hg19_rRNAtRNAchrM -u \
-o outPutLinks accepted_filtered.bam
重要参数解释:
-p/--num-threads:设置线程数,用于多核计算。-o/--output-dir:输出文件夹。-g/--GTF-guide:使用参考转录组注释文件指导组装过程,建议添加。加入这个参数和对应的注释文件,可以有效提高cufflinks转录本组装。输出的转录组是传入注释文件和cufflinks自行组装的联合。具体解释如下图[[Roberts et.al., 2011]](#Roberts et.al., 2011):
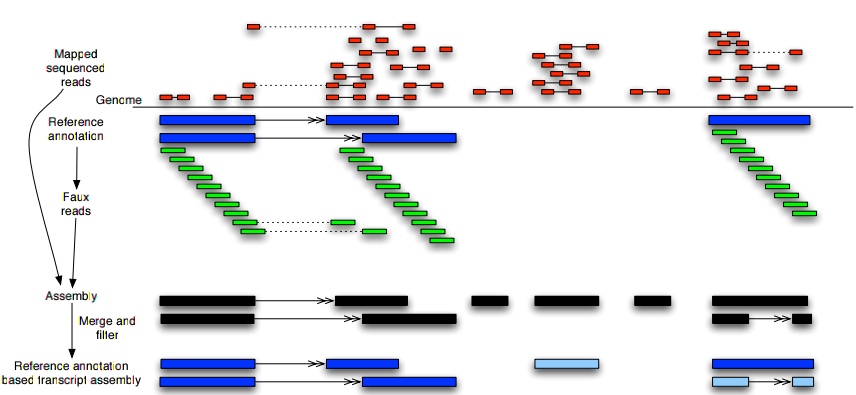
-G/--GTF:只对传入的转录组注释文件进行表达量估计,即生成的结果如genes.fpkm_tracking只包括转录组注释文件,而不包括cufflinks自己可能的组装。如果RNA-seq分析的目的是为了获得差异表达基因,而不是寻找新的转录本,建议添加,以加快运行速度。-b/--frag-bias-correct:提供一个multifasta文件,可以提高转录丰度的准确度,建议添加。-M/–mask-file:需要去除的转录本,比如tRNA、rRNA和线粒体转录本,建议添加。在RNA-seq数据中,这些转录本量非常高。cufflinks提示剔除这些转录本,有助于提高转录量估计的准确程度。tRNA、rRNA和线粒体基因组注释信息提取,参考UCSC Table下载注释文件。-u/--multi-read-correct:处理map到基因组多个位置的reads,建议添加。对于TopHat,建议过滤掉这部分reads,过滤方法见过滤TopHat分析双端测序的输出。按照上述方法过滤这部分reads后,则不需要加这个参数。--library-type:设定测序方法,默认为fr-unstranded,可以用于典型的Illumina双端(paired-end)测序。
2.2 cuffmerge 快速运行
$ cuffmerge -p 8 -g hg19USCS_ensembl.gtf -s hg19.fa -o mergeFile assemblies.txt
重要参数解释:
-p/--num-threads:设置线程数,用于多核计算。-o/--output-dir:输出文件夹。g/--ref-gtf:参考转录组,建议添加。g/--ref-gtf:参考基因组序列,建议添加。assemblies.txt:是一个文件,写入需要组装转录本的路径。
2.3 cuffquant 快速运行
$ cuffquant -p 14 -o quantOut -b hg19.fa \
-M maskfile.gtf -u \
merged.gtf h1.bam
重要参数解释:
-p/--num-threads:设置线程数,用于多核计算。-o/--output-dir:输出文件夹。-b/--frag-bias-correct:参考基因组序列,建议添加。-M/--mask-file:过滤序列GTF文件,放入这个文件中的序列都不会被计算,建议添加。-u/--multi-read-correct:处理map到基因组多个位置的reads,建议添加。
2.4 cuffdiff快速运行
# cuffdiff可以输入cuffquant生成的cxb文件或者原始的bam文件
$ cuffdiff -p 14 -o diffOut -b hg19.fa \
-M maskfile.gtf -u \
-L P,R merged.gtf h1.cxb,h2.cxb,h5.cxb h2.cxb,h4.cxb,h6.cxb
重要参数解释:
-p/--num-threads:设置线程数,用于多核计算。-o/--output-dir:输出文件夹。-b/--frag-bias-correct:参考基因组序列,建议添加。-M/--mask-file:过滤序列GTF文件,放入这个文件中的序列都不会被计算,建议添加。-u/--multi-read-correct:处理map到基因组多个位置的reads,建议添加。-L/--labels:指定样品组名
3. CummeRbund
个人感觉CummeRbund不好用,不做介绍和讨论。
参考资料
Roberts A, Pimentel H, Trapnell C, Pachter L: Identification of novel transcripts in annotated genomes using RNA-Seq. Bioinformatics. 2011, 27(17):2325-9. pdf
更新记录
2015年6月10日